Alte Genome geben Einblick in die genetische Geschichte der zweiten Pestpandemie
Die Analyse von 34 Pestgenomen aus der Zeit des Schwarzen Todes und nachfolgender Epidemien zwischen dem 14. und 17. Jahrhundert in Europa zeigt, wie sich das Bakterium nach einem einzigen Eintrag nach Europa diversifizierte.
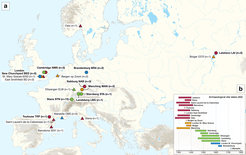
Die Karte zeigt die archäologischen Stätten der neu sequenzierten (Kreise) und bereits publizierter Pestgenome (Dreiecke), in zeitlicher Staffelung gefärbt.
Die zweite Pestpandemie begann Mitte des 14. Jahrhunderts mit dem "Schwarzen Tod" und dauerte bis ins 18. Jahrhundert an. Den verheerenden Ausbrüchen in ganz Europa und in angrenzenden Regionen fielen bis zu 60 Prozent der Bevölkerung zum Opfer. Aber woher kam dieser Stamm des Pesterregers Yersinia pestis? Und wie hat sich das Bakterium, nachdem es Europa erreicht hatte, weiterentwickelt und ausgebreitet?
Y.-pestis-Stämme der zweiten Pandemie gehen vermutlich alle auf einen Urahnen zurück
Da nur wenige Daten zu frühen Ausbrüchen der Pest vorliegen und ein Mangel an veröffentlichten genetischen Daten historischer Pestgenome besteht, ist trotz der Allgegenwart des Schwarzen Todes in historischen Texten und im allgemeinen Bewusstsein nicht klar, von wo und über welche Route das Y. pestis-Bakterium zu dieser Zeit nach Europa gelangte und wie es sich über den Kontinent ausbreitete. In der aktuellen Studie rekonstruierten die Wissenschaftlerinnen und Wissenschaftler das Pestgenom aus den Zähnen von 34 Pestopfern, darunter zwei aus Laishevo, in der Wolga-Region Russlands. Bei der Analyse dieser Pestgenome gelang es ihnen, einen Bakterienstamm zu identifizieren, von dem alle weiteren Bakterienstämme der zweiten Pandemie abstammen.
Darüber hinaus beobachtete das Team bei den Proben aus der Zeit des Schwarzen Todes das Fehlen genetischer Vielfalt. "Diese Ergebnisse deuten darauf hin, dass das Yersinia pestis von Osten her nach Europa gelangte", erklärt Ko-Erstautorin Maria Spyrou vom Jenaer Max-Planck-Institut für Menschheitsgeschichte. "Es ist jedoch möglich", fährt sie fort, "dass zukünftig weitere Interpretationen möglich werden, wenn zum Beispiel eine bislang unbekannte genetische Vielfalt des Bakteriums in West-Eurasien entdeckt würde."

Der Fortbestand und die Entwicklung des Y.-pestis-Bakteriums in Europa
Obwohl die Studie zeigt, dass der europaweite Schwarze Tod wahrscheinlich durch einen einzigen Stamm ausgelöst wurde, belegt die Analyse von Genomen aus der späteren Pandemie die Entstehung einer Linie mit einer höheren genetischen Vielfalt. "In der späteren Phase der zweiten Pandemie sehen wir die Entwicklung zweier Zweige in Europa, was darauf hindeutet, dass das Pestbakterium in verschiedenen lokalen Reservoiren überlebte", sagt Ko-Erstautor Marcel Keller vom Max-Planck-Institut für Menschheitsgeschichte. „Moderne Nachfahren dieser Linie wurden jedoch bislang nicht gefunden, was auf ein Aussterben dieser Reservoire hindeutet.“
Das Team entdeckte außerdem in Genomen eines Zweigs den Verlust eines Erbgutteils, darunter zwei Gene, die mit der Virulenz des Bakteriums in Verbindung stehen, also wie gefährlich bzw. ansteckend der Erreger ist. Interessanterweise wurde in Genomen der späten ersten Pandemie eine Deletion der gleichen Region festgestellt. "Angesichts der Tatsache, dass diese Deletion in Linien der ersten und zweiten Pandemie auftritt, die beide inzwischen ausgestorben sind, wäre es wichtig in zukünftigen Studien zu klären, wie sich diese Gene auf das Überleben des Bakteriums in Menschen und Flöhen auswirken", sagt Kirsten Bos, Forschungsgruppenleiterin am Max-Planck-Institut für Menschheitsgeschichte.

Die aktuelle Studie eröffnet neue Perspektiven auf den Beginn und Verlauf der zweiten Pandemie und ergänzt die Datenbank der veröffentlichten historischen Pestgenome signifikant. "Wir konnten zeigen, dass eine umfassende Analyse alter Y.-pestis-Genome einzigartige Einblicke in die Mikroevolution eines Erregers über einen Zeitraum von mehreren hundert Jahren liefern kann", sagt Johannes Krause, Direktor der Abteilung für Archäogenetik am Max-Planck-Institut für Menschheitsgeschichte und Leiter der Studie. "In Zukunft wird die Integration dieser Daten in die Modellierung von Krankheitsausbrüchen, zusammen mit Daten aus anderen Disziplinen wie der Klimaforschung, Epidemiologie und Geschichte, für ein besseres Verständnis der zweiten Pandemie von Bedeutung sein."


